Pada masalah klasifikasi data yang tidak seimbang ada beberapa teknik yang dapat dilakukan. Pertama adalah dengan membuat algoritma yang “super” agar dapat melakukan prediksi yang akurat. Sebagaimana yang sering kita baca di paper-paper yang telah ada, bahwa algoritma-algoritma klasifikasi yang telah ada “tidak mampu” untuk menyelesaikan kasus ketika data tidak seimbang (suatu class berjumlah lebih besaaaaar dibanding class lainnya). Untuk mengembalikan kemampuan tersebut, maka perlu menyeimbangkan data terlebih dahulu. Setelah data seimbang maka algoritma yang telah ada saat ini dapat digunakan kembali dengan harapan prediksinya kembali akurat.
Teknik penyeimbangan data tersebut terbagi 2, yaitu:
- Undersampling yaitu mengurangi data, biasanya mengurangi data class yang lebih banyak/mayoritas.
- Oversampling yaitu menambah data, biasanya menambah data class minoritas.
Nah posting ini bertujuan untuk melihat apa yang terjadi terhadap data setelah dilakukan proses penyeimbangan. Efek yang ingin saya lihat adalah efek sebaran data pada data space, sehingga akan coba saya visualisasikan dalam bentuk grafik. Teknik yang akan digunakan adalah SMOTE dan ROSE. Dataset yang digunakan adalah Iris, Yeast dan Abalone. Kedua data terakhir diambil dari UCI Machine Learning Repository.
{SMOTE}
Berikut adalah kode yang digunakan untuk melakukan pengujian.
#library
library(pca3d)
library(DMwR)
#iris - start
#-----------------------------------
#membuat data iris menjadi unbalance
main_data = iris
main_data[which(main_data$Species == "versicolor"),5] = "virginica"
main_data[,5] = factor(main_data[,5])
main_data = rbind(main_data[which(main_data$Species == "virginica"),], main_data[which(main_data$Species == "setosa"),][1:20,])
#melihat sebaran data - awal
main_data.pca = prcomp(main_data[,-5])
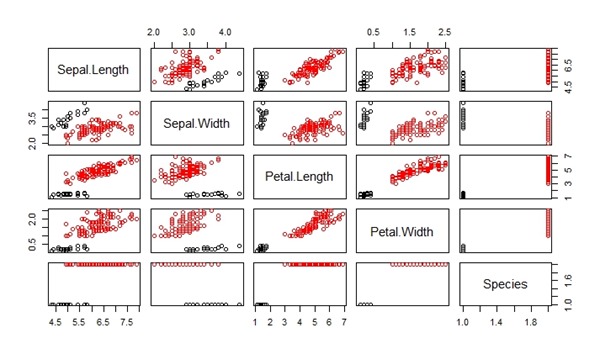
plot(main_data, col = main_data[,5])
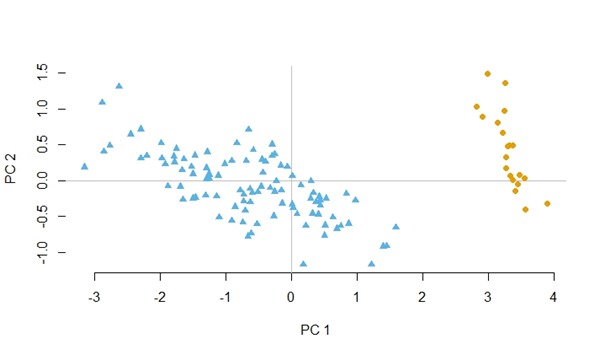
pca2d(main_data.pca, group = main_data[,5])
#undersampling & oversampling with SMOTE
main_data = SMOTE(Species ~ ., main_data, perc.over = 100, perc.under = 200, k = 5)
#melihat sebaran data - setelah sampling
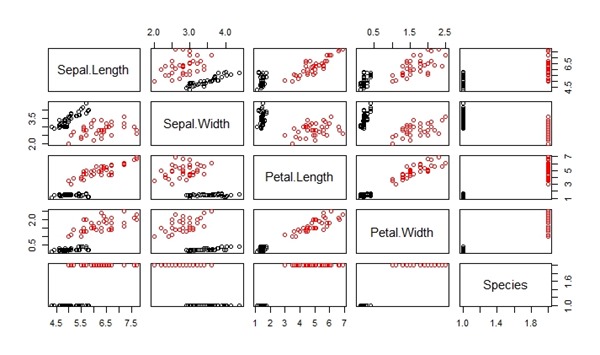
plot(main_data, col = main_data[,5])
main_data.pca = prcomp(main_data[,-5])
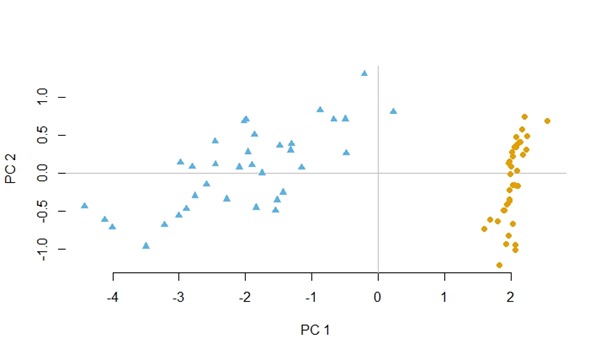
pca2d(main_data.pca, group = main_data[,5])
#-----------------------------------
#iris - start
#yeast - start
#-----------------------------------
#data yeast belum seimbang
yeast = read.csv("yeast2class.csv")
#melihat sebaran data - awal
plot(yeast, col = yeast[,9])
yeast.pca = prcomp(yeast[,-9])
pca2d(yeast.pca, group = yeast[,9])
#undersampling & oversampling with SMOTE
yeast = SMOTE(V9 ~ ., yeast, perc.over = 100, perc.under = 200, k = 5)
#melihat sebaran data - setelah sampling
plot(yeast, col = yeast[,9])
yeast.pca = prcomp(yeast[,-9])
pca2d(yeast.pca, group = yeast[,9])
#-----------------------------------
#yeast - end
#abalone - start
#-----------------------------------
#data abalone sebelum seimbang
abalone = read.csv("abalone2class.csv")
#melihat sebaran data - awal
plot(abalone, col = abalone[,8])
abalone.pca = prcomp(abalone[,-8])
pca2d(abalone.pca, group = abalone[,8])
#undersampling & oversampling with SMOTE
abalone = SMOTE(V8 ~ ., abalone, perc.over = 100, perc.under = 200, k = 5)
#melihat sebaran data - setelah sampling
plot(abalone, col = abalone[,8])
abalone.pca = prcomp(abalone[,-8])
pca2d(abalone.pca, group = abalone[,8])
#-----------------------------------
#abalone - end
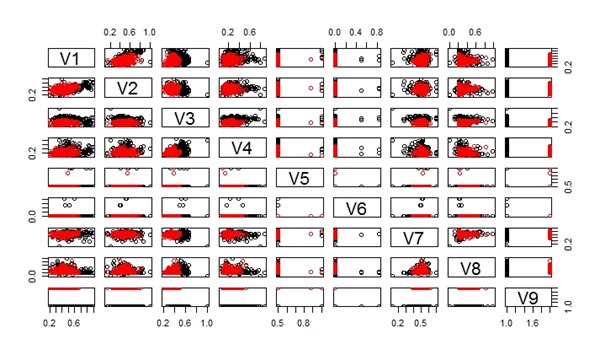
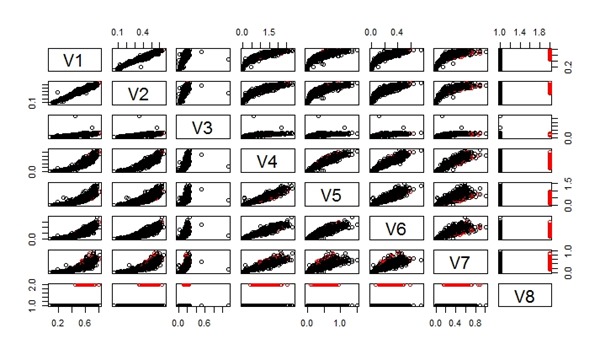
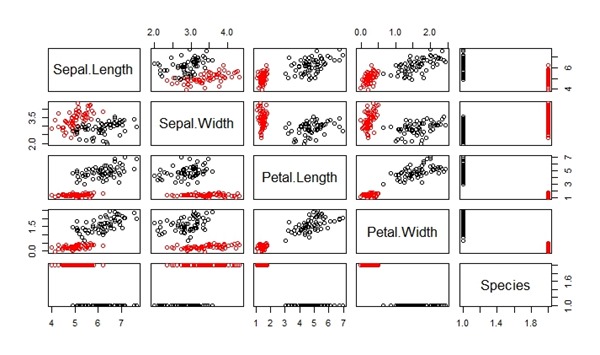
Berikut adalah hasil dari kode di atas. Dibawah ini adalah efek pada data Iris dengan membandingkan antara sebelum dan sesudah dilakukan penyeimbangan data.
Sebelum dilakukan penyeimbangan data.
Setelah dilakuan penyeimbangan data.
Sebelum dilakukan penyeimbangan data.
Setelah dilakukan penyeimbangan data.
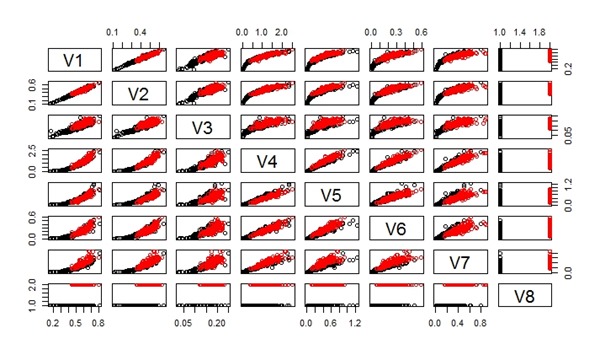
Berikut ini adalah perbandingan keadaan sebelum dan sesudah melakukan penyeimbangan data pada dataset Yeast.
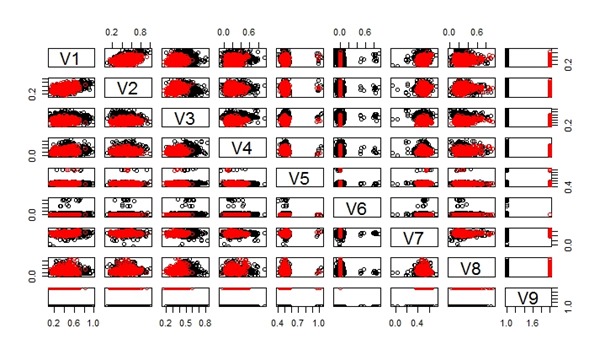
Data yeast awal.
Data yeast setelah dilakukan penyeimbangan.
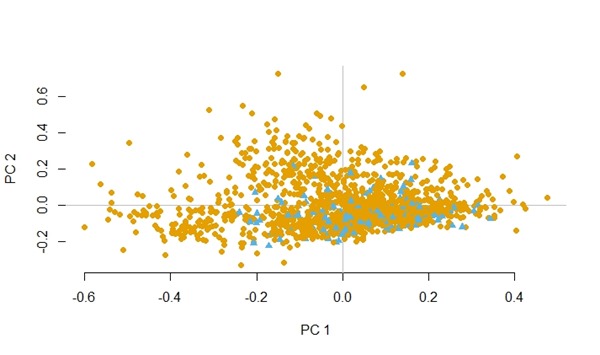
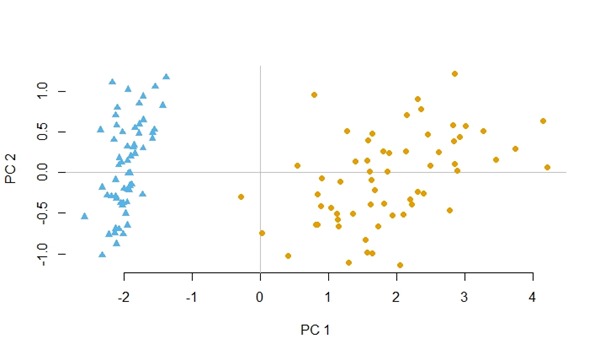
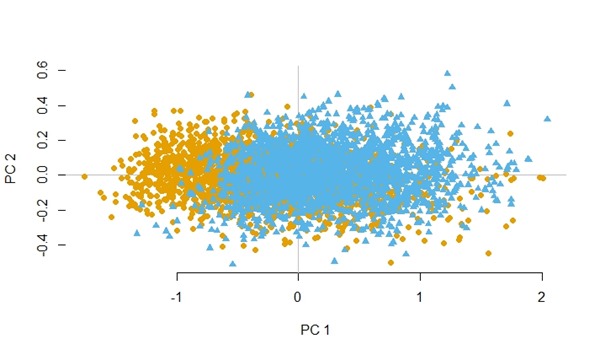
Visualisasi data yeast dengan PCA sebelum diseimbangkan.
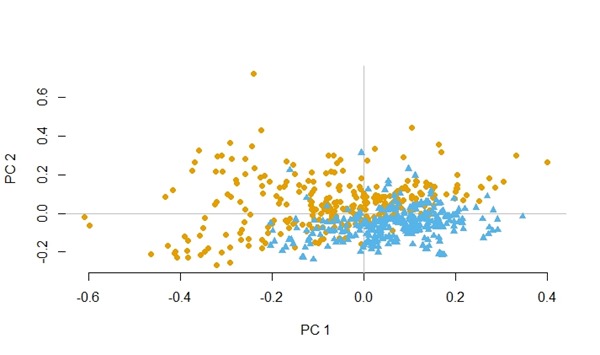
Visualisasi data yeast dengan PCA setelah diseimbangkan.
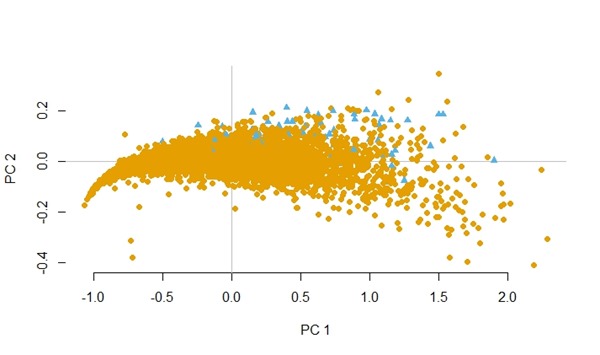
Selanjutnya adalah efek penyeimbangan data dengan SMOTE pada dataset Abalone.
data awal abalone.
Data abalone setelah diseimbangkan.
Visualisasi data abalone dengan PCA sebelum diseimbangkan.
Visualisasi data abalon dengan PCA setelah diseimbangkan.
{ROSE}
Berikut ini adalah kode yang digunakan untuk menyeimbangkan data dengan teknik ROSE.
#library
library(pca3d)
library(ROSE)
#iris - start
#-----------------------------------
#membuat data iris menjadi unbalance
main_data = iris
main_data[which(main_data$Species == "versicolor"),5] = "virginica"
main_data[,5] = factor(main_data[,5])
main_data = rbind(main_data[which(main_data$Species == "virginica"),], main_data[which(main_data$Species == "setosa"),][1:20,])
#melihat sebaran data - awal
main_data.pca = prcomp(main_data[,-5])
plot(main_data, col = main_data[,5])
pca2d(main_data.pca, group = main_data[,5])
#undersampling & oversampling with SMOTE
main_data = ROSE(Species ~ ., main_data)
main_data = main_data$data
main_data = rbind(main_data[which(main_data$Species == "setosa"),],main_data[which(main_data$Species == "virginica"),])
#melihat sebaran data - setelah sampling
plot(main_data, col = main_data[,5])
main_data.pca = prcomp(main_data[,-5])
pca2d(main_data.pca, group = main_data[,5])
#-----------------------------------
#iris - start
#yeast - start
#-----------------------------------
#data yeast belum seimbang
yeast = read.csv("yeast2class.csv")
#melihat sebaran data - awal
plot(yeast, col = yeast[,9])
yeast.pca = prcomp(yeast[,-9])
pca2d(yeast.pca, group = yeast[,9])
#undersampling & oversampling with SMOTE
yeast = ROSE(V9 ~ ., yeast)
yeast = yeast$data
#melihat sebaran data - setelah sampling
plot(yeast, col = yeast[,9])
yeast.pca = prcomp(yeast[,-9])
pca2d(yeast.pca, group = yeast[,9])
#-----------------------------------
#yeast - end
#abalone - start
#-----------------------------------
#data abalone sebelum seimbang
abalone = read.csv("abalone2class.csv")
#melihat sebaran data - awal
plot(abalone, col = abalone[,8])
abalone.pca = prcomp(abalone[,-8])
pca2d(abalone.pca, group = abalone[,8])
#undersampling & oversampling with SMOTE
abalone = ROSE(V8 ~ ., abalone)
abalone = abalone$data
#melihat sebaran data - setelah sampling
plot(abalone, col = abalone[,8])
abalone.pca = prcomp(abalone[,-8])
pca2d(abalone.pca, group = abalone[,8])
#-----------------------------------
#abalone - end
Hasilnya adalah gambar-gambar di bawah ini.
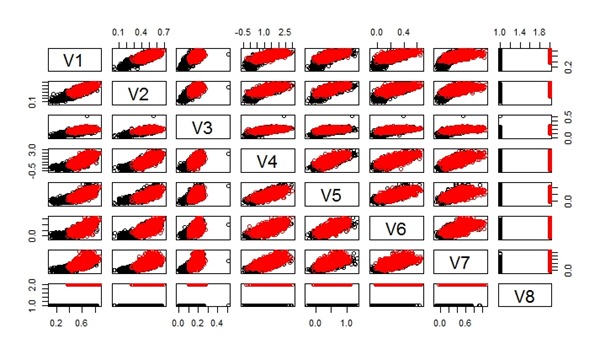
Dataset iris setelah diseimbangkan dengan teknik ROSE.
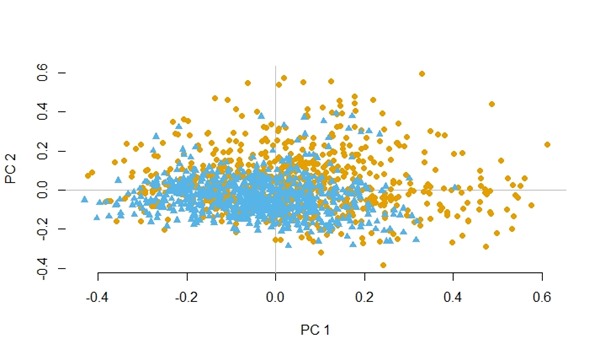
Visualisasi setelah diseimbangkan (hasil PCA).
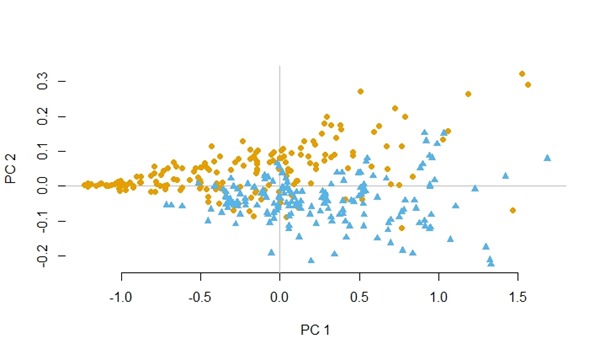
Berikut ini adalah dataset yeast setelah diseimbangkan dengan teknik ini.
Dan di bawah ini adalah dataset abalone setelah diseimbangkan.
{Kesimpulan}
Dari kedua teknik yang dicoba di atas, ada beberapa hal yang menjadi catatan pribadi saya, yaitu:
- Keduanya teknik di atas, SMOTE dan ROSE, mempunyai kesamaan yaitu hanya menambah dan/atau mengurangi instance agar jumlahnya seimbang. Tetapi terlihat tidak mempengaruhi luas area “kekuasaan” masing-masing class.
- Terlihat proses penambahan data/atau pengurangan data lebih berfokus melakukan penyeimbangan kepada daerah overlapping. Hal ini sepertinya agar antara (yang semula) class minoritas dan (yang semula) class mayoritas dapat bertarung dengan kekuatan yang sama di daerah overlapping.
- Tetapi jika ada pengurangan maka pengurangan akan berfokus kepada daerah yang tidak terjadi overlapping. Karena tidak terjadi pertarungan penting di daerah ini, maka tidak daerah tersebut tidak perlu diperhatikan atau dijaga.
Dari kedua kesimpulan di atas, maka ada beberapa catatan pribadi terkait dengan riset yang sedang dikerjakan, yaitu:
- rahasia.
- rahasia.
Untuk memperbaiki kesalahan pada point pertama dengan cara point kedua tanpa harus memodifikasi jumlah instance setiap class, maka saya merencanakan studi berikut ini:
- rahasia.
- rahasia.
Doakan saya ya ![]()